De la microbiologie pour ceux qui ont les crocs
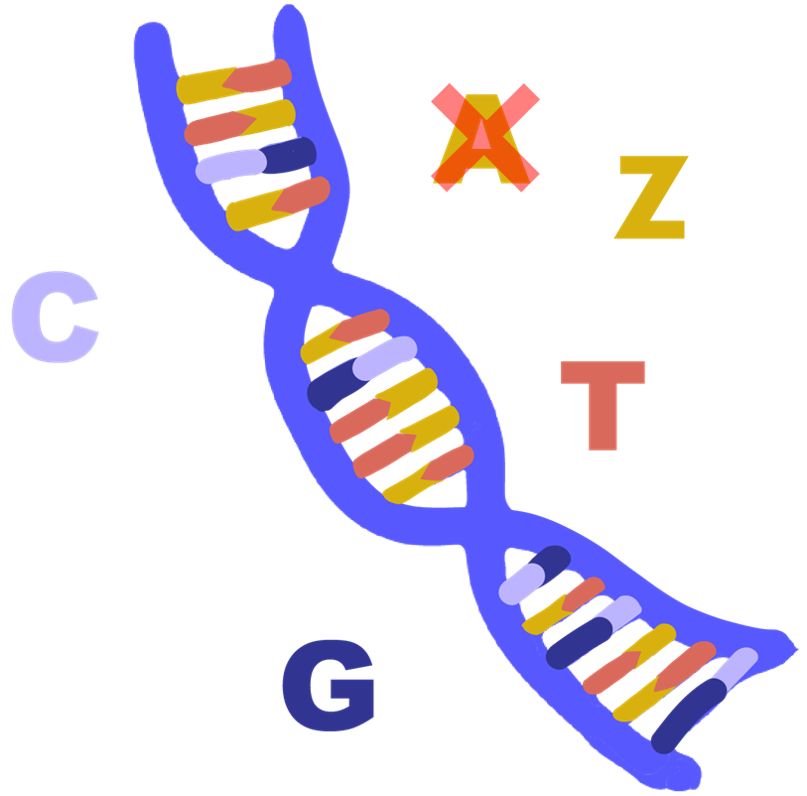
Une nouvelle lettre dans l’alphabet génétique.
Tout l’ADN est fabriqué en utilisant le même alphabet. Des virus aux êtres humains, nous utilisons tous le code à quatre lettres: A, C, T, G. Ces quatre nucléotides (Adénine, Cytosine, Thymine et Guanine) constituent le code génétique de tous les organismes.
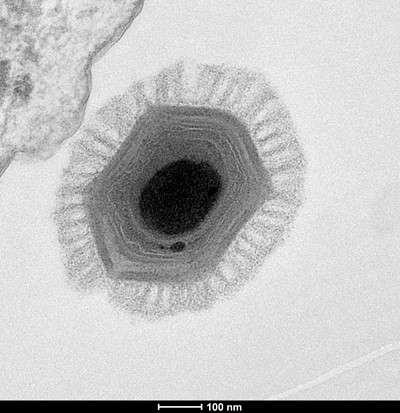
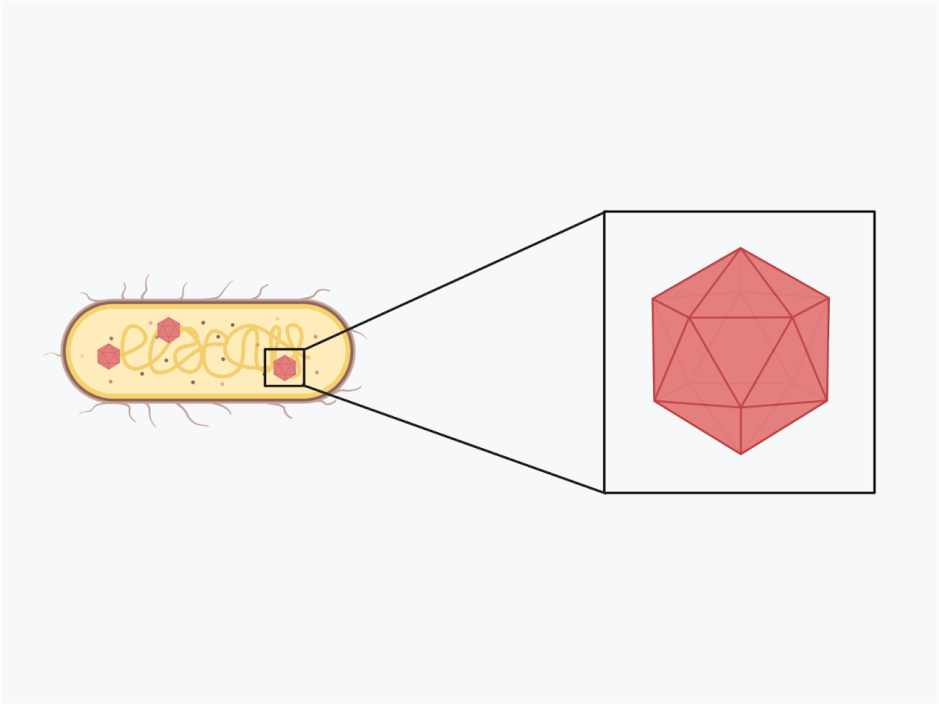
Cependant en 1977, une anomalie a été trouvée. Un bactériophage (S-2L) infectant des cyanobactéries s’est avéré ne pas avoir la lettre A mais un nucléotide étroitement apparenté: la diaminopurine ou Z. Les modifications de l’ADN ne sont pas rares, mais ici, la nouvelle lettre a complètement remplacé A dans l’appariement d’ADN du phage avec sa base complémentaire T.

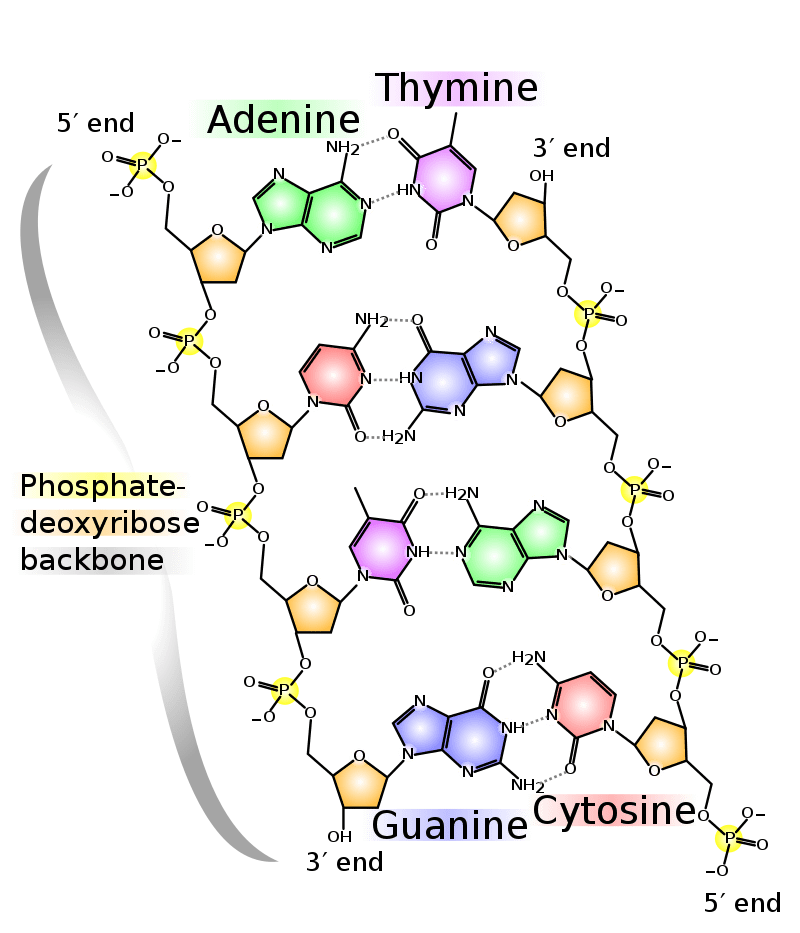
Depuis, d’autres études se sont penchées sur cette nouvelle lettre d’ADN en termes de voies de synthèse et de structure au sein de l’ADN, mais avec des résultats limités. L’ADN (acide désoxyribonucléique) est composé d’un squelette constitué d’une alternance de groupes phosphate P-O qui confèrent à l’ADN ses propriétés acides, et d’un sucre appelé 2-désoxyribose: un pentose. La partie nucléique du nom provient de notre code à quatre lettres, les nucléotides. Les nucléotides maintiennent les deux brins en double hélice par une force d’attraction constituée de liaisons hydrogène. La paire A-T forme deux liaisons hydrogène tandis que C-G en forme trois comme on le voit sur la figure.
En 1998, une étude a montré que la paire Z-T dans le phage lytique S-2L formait trois liaisons hydrogène, modifiant ainsi la structure 3-D de l’ADN (plus d’informations sur les phages lytiques ici).

Cependant, ce n’est qu’en avril 2021 que plusieurs groupes scientifiques français et chinois ont trouvé la même «anomalie» chez d’autres bactériophages, et ont pu mieux caractériser les mécanismes de la nouvelle voie de biosynthèse ainsi que ses implications (REF1, REF2, REF3).
Les groupes de recherche de Yan Zhou et de Dona Sleiman ont montré que la lettre Z était utilisée par d’autres phages, tandis que la troisième étude se concentrait principalement sur un phage de la bactérie Vibrio: Phi-VC8. En étudiant comment le nouveau nucléotide est utilisé dans les différents organismes, les groupes de Zhou et de Sleiman ont examiné plus en détail les voies de synthèse impliquées par son enzyme PurZ.
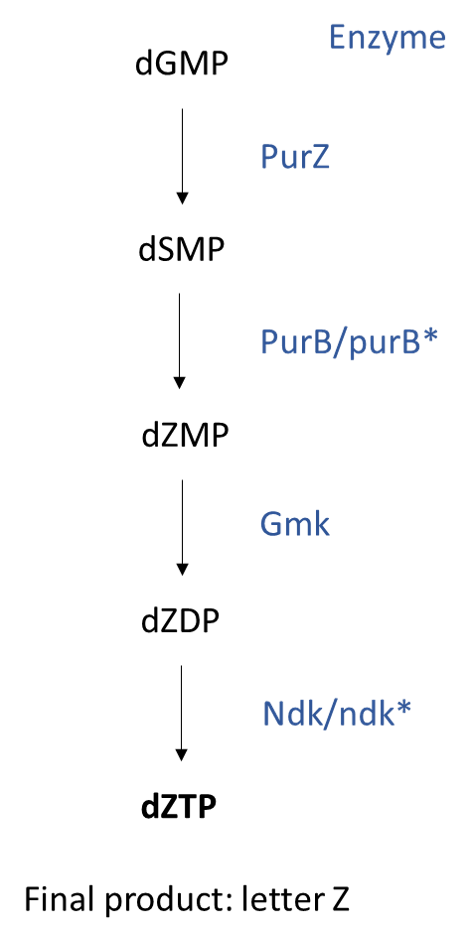
En utilisant plusieurs mutants, Sleiman et ses collègues ont montré quelles enzymes étaient essentielles pour synthétiser le nouveau nucléotide et ont pu cartographier sa synthèse à partir du dGMP (guanine monophosphate) jusqu’au dZTP (Z triphosphate). Ils ont montré que les mutants purB* avaient une efficacité d’infection plus limitée tandis que les mutants ndk * n’infectaient pas du tout les cellules. Ces résultats mettent en évidence que ces gènes sont nécessaires pour la synthèse du dZTP et sans eux, les phages ne peuvent pas se répliquer.
La dernière étude de Valerie Pezo et de ses collègues s’est d’avantage concentrée sur la machinerie de l’ADN, en particulier l’ADN polymérase (plus d’informations sur la machinerie de l’ADN ici). Les ADN polymérases sont utilisées pour répliquer l’ADN : elles se lient à chaque simple brin et lisent la séquence dans le sens 5’-3 ’. Au fur et à mesure qu’elle lit, elle ajoute les nucléotides complémentaires (T pour A, C pour G et vice versa) de leur forme NTP libre (nucléotide triphosphate, TTP, ATP, GTP ou CTP).

Wikicommons
La question est donc si ces phages ont une ADN polymérase différente pour utiliser le ZTP lors de la réplication de leur ADN? L’étude a montré que l’ADN polymérase trouvée dans les organismes utilisant des ZTP était 30 à 90 fois plus efficace dans l’utilisation des ZTP au lieu des ATP. Inversement, l’ADN polymérase d’E. coli était environ deux fois plus efficace pour insérer l’ATP lorsque l’ATP et le ZTP étaient tous deux présents dans la cellule. Par conséquent, il semble que ces organismes disposent d’une ADN polymérase optimisée pour utiliser le nouvel alphabet.
Quelles sont les implications? Pourquoi ces virus ont-ils évolué de cette façon? Zhou et ses collègues pensent que cela donne un avantage évolutif aux phages en évitant les défenses de l’hôte contre les attaques par enzymes de restriction. La compréhension de ce nouvel alphabet pourrait avoir un impact majeur sur différents domaines tels que la fabrication de nanostructures à partir d’ADN, également appelé ADN origami, ou dans le stockage de données basé sur l’ADN. Enfin, ce nouvel alphabet pourrait être utilisé pour concevoir des phages afin d’augmenter la gamme de cellules hôtes et l’efficacité de la phagothérapie, ainsi que la conservation des aliments parmi d’autres utilisations potentielles (en savoir plus sur la phagothérapie ici).
Peut-être que l’alphabet ADN que nous connaissons aujourd’hui s’étendra à mesure que nous étudierons de plus en plus d’organismes, en particulier les virus. Bien que nous ayons essayé de créer nous-mêmes de nouvelles lettres, il semble que la nature ait déjà fait le travail à notre place.
Articles:
Featured image: Modifiée de: https://innovativegenomics.org/glossary/dna/