De la microbiologie pour ceux qui ont les crocs
Les origines procaryotiques de notre plus grande méthode de modification génétique
CRISPR est aujourd’hui la référence en matière de modification génétique. Elle est plus rapide, moins chère et plus efficace que les méthodes concurrentes. Les scientifiques ont découvert que CRISPR peut être utilisé pour modifier les génomes des plantes et des animaux (y compris les humains) [1]. La “thérapie génique” pourrait, par exemple, traiter les maladies héréditaires ou lutter contre le vieillissement. Par exemple, des chercheurs chinois ont utilisé CRISPR pour modifier le gène CCR5 chez des jumeaux [2]. CCR5 permet l’infection par le VIH, la modification de ce gène pourrait protéger contre l’infection virale. Cet exemple montre les applications modernes de CRISPR. Mais d’où vient le CRISPR ? Contrairement à son utilisation dans les cellules humaines, cet outil moléculaire est né chez les prokayotes.
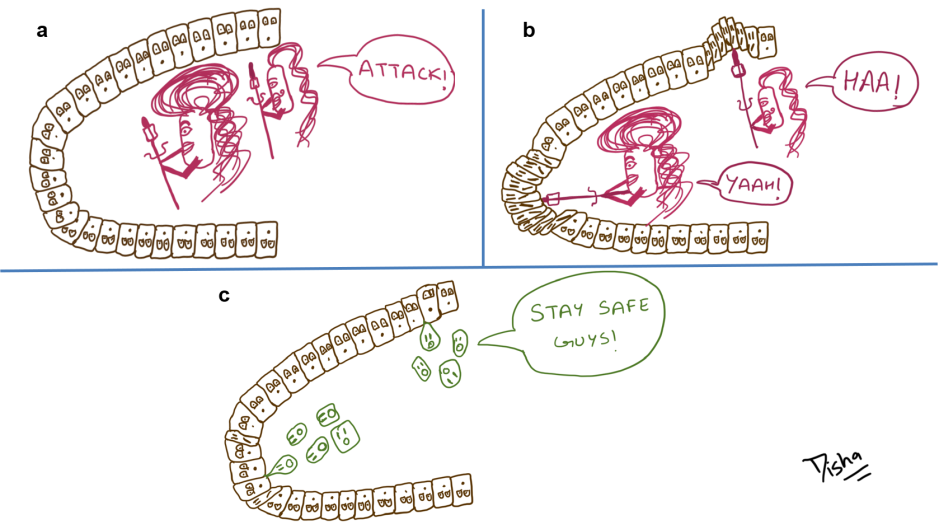
CRISPR est un “système immunitaire” adaptatif des procaryotes. Les bactéries et les archées sont constamment visées par des “phages”, un type de virus. L’un des outils utilisés pour la protection est le CRISPR – une abréviation anglaise pour Clustered Regularly Interspaced Short Palindromic Repeats = « Courtes répétitions palindromiques groupées et régulièrement espacées ». Une région spécifique du génome procaryote, le locus CRISPR, contient de nombreuses copies de la même séquence. Ces séquences répétées sont entrecoupées de morceaux d’ADN variables appelés spacers (figure 1A) [3]. Ces spacers sont des morceaux d’ADN provenant d’un virus qui a été vaincu par le procaryote dans le passé. Tout comme notre propre système immunitaire stocke des informations provenant d’envahisseurs précédents, CRISPR constitue la “mémoire moléculaire” des attaques virales. L’avantage d’une telle mémoire est que le matériel génétique d’un visiteur indésirable peut être rapidement reconnu et détruit.

L’existence de séquences CRISPR a été signalée pour la première fois en 1993 par Francisco Mojica [1]. La fonction biologique de ce locus était alors inconnue. Près de vingt ans plus tard, des chercheurs de la société Danisco à Copenhague ont démontré la fonction de CRISPR : un système immunitaire contre les phages [3]. L’industrie laitière a beaucoup investi dans la compréhension du système immunitaire bactérien, car de nombreux produits laitiers pouvaient être gravement affectés par les phages. Rodolphe Barrangou et Phillipe Horvath, de Danisco, ont montré expérimentalement que les séquences CRISPR immunisent une bactérie contre les phages qui ont des séquences homologues (les mêmes) dans leur propre génome [3].
Pour leurs recherches, Barrangou et Horvath ont utilisé Streptococcus thermophilus, une bactérie lactique largement utilisée dans l’industrie laitière. Ils ont constaté que les souches de bactéries de cette espèce qui présentaient une résistance accrue aux phages avaient un plus grand nombre de spacers distincts dans leur locus CRISPR. Cela indique que CRISPR a quelque chose à voir avec l’immunité [3].
Comment S. thermophilus obtient-il les spacers dans son génome ? L’étude a montré que les loci CRISPR sont une partie dynamique du génome, qui change rapidement pendant les attaques de phages [3]. Les Danois ont étudié cette question en exposant une souche bactérienne dotée d’un faible système immunitaire au “phage #858”, qu’ils ont trouvé dans un pot de yaourt. En cultivant les bactéries survivantes, les chercheurs ont créé leur propre S. thermophilus résistant à 858. Tous les survivants avaient des morceaux d’ADN supplémentaires dans le locus CRISPR. Ces morceaux étaient tous des fragments de l’ADN du phage 858. L’homologie entre le spacer et l’ADN infectieux semble donc être déterminante pour l’immunité de la bactérie.
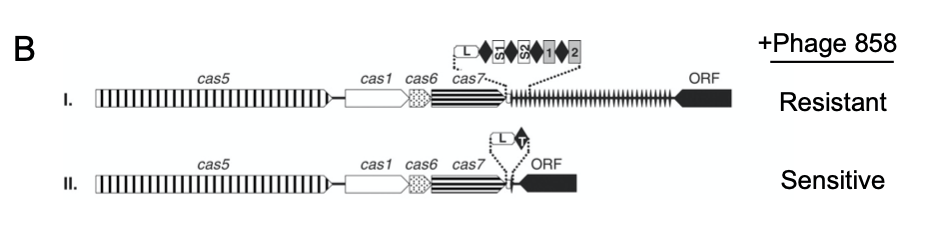
Pour vérifier si cette théorie était correcte, Barrangou et Horvath ont coupé les spacers d’une souche bactérienne résistante au 858 et les ont collés au même endroit dans une souche sensible au 858 (figure 1B). Résultat : la souche modifiée s’est avérée résistante au phage 858 ! Et ça fonctionne également dans l’autre sens : en retirant le spacer qui présentait une homologie avec le phage 858, une bactérie résistante pouvait soudainement être infectée par le phage 858. Les spacers offrent donc une immunité très ciblée [3].
Les spacers ne sont pas les seuls éléments du système immunitaire CRISPR. Les gènes cas, qui se trouvent juste à côté des CRISPR, sont également importants pour l’immunité virale. Depuis la découverte de Barrangou et Horvath, des études pionnières ont révélé les rôles de ces gènes [1]. En comprenant la vision globale de CRISPR, on a pu en faire un outil de laboratoire. Ainsi, un système immunitaire bactérien a entraîné une explosion de nouvelles applications en biotechnologie et en médecine !
Literature Cited
- Lander, E.S. (2016). The Heroes of CRISPR. Cell 164, 18-28.
- Raposo, V.L. (2019). The First Chinese Edited Babies: A Leap of Faith in Science. JBRA Assist Reprod. 23, 197-199.
- Barrangou, R., Fremaux, C., Deveau, H., Richards, M., Boyaval, P., Moineau, S., Romero, D.A., and Horvath, P. (2007). CRISPR provides acquired resistance against viruses in prokaryotes. Science 315, 1709–1712.
Featured image: https://i1.wp.com/foodcrumbles.com/wp-content/uploads/2018/07/Home-made-yogurt-1-2.jpg?fit=750%2C500&ssl=1