De la microbiologie pour ceux qui ont les crocs
Une connexion profondément enracinée
Chaque espèce végétale est associée à une collection unique de micro-organismes. Les communautés microbiennes autour des racines varient un peu en fonction du type de sol, mais sont assez robustes. Nous savons quels organismes finissent par entretenir une relation intime, mais qui fait le premier pas ? Une plante attire-t-elle les bactéries qui lui plaisent, ou une bactérie s’attaque-t-elle aux plantes qui lui plaisent ? Katrin Wippel et Ke Tao (ainsi qu’une équipe internationale de spécialistes des plantes) ont décidé d’analyser la première étincelle.
Les scientifiques ont étudié deux espèces de plantes pour mettre en évidence les différences dans leur préférence pour les bactéries : Arabidopsis thaliana (une cousine du chou) et Lotus japonica (une cousine du haricot). Les chercheurs ont d’abord déterminé quelles bactéries se trouvent autour des racines des deux plantes dans leur milieu naturel en utilisant le séquençage de l’ARN 16S. Ils ont trouvé des centaines d’espèces bactériennes autour des racines des deux plantes.
Comme il est difficile de travailler avec des centaines d’espèces bactériennes, les chercheurs ont créé un microbiome simplifié pour les deux espèces végétales : une communauté synthétique (SynCom), voir Figure 1 ci-dessous. 16 espèces bactériennes ont été choisies pour A. thaliana, et 16 pour L. japonica. Wippel et Tao ont choisi ces espèces de manière à ce que chaque espèce bactérienne d’un SynCom ait un parent proche dans l’autre (partageant la même famille bactérienne). De cette façon, les SynCom de A. thaliana et de L. japonica étaient très similaires. Pouvaient-ils tromper A. thaliana pour qu’elle travaille avec les bactéries de L. japonica, ou vice-versa ?
Pour essayer de tromper les espèces végétales, les chercheurs ont mélangé les deux SynComs pour obtenir un total de 32 espèces bactériennes, mais toujours seulement 16 familles. Ils ont ensuite inoculé ce mélange à A. thaliana et L. japonica. Après 5 semaines, les deux plantes ont été retrouvées avec seulement leur communauté respective de 16 bactéries enrichies sur leurs racines ! Les plantes n’ont pas été dupes, quelque chose dans l’interaction semble donc être très spécifique.
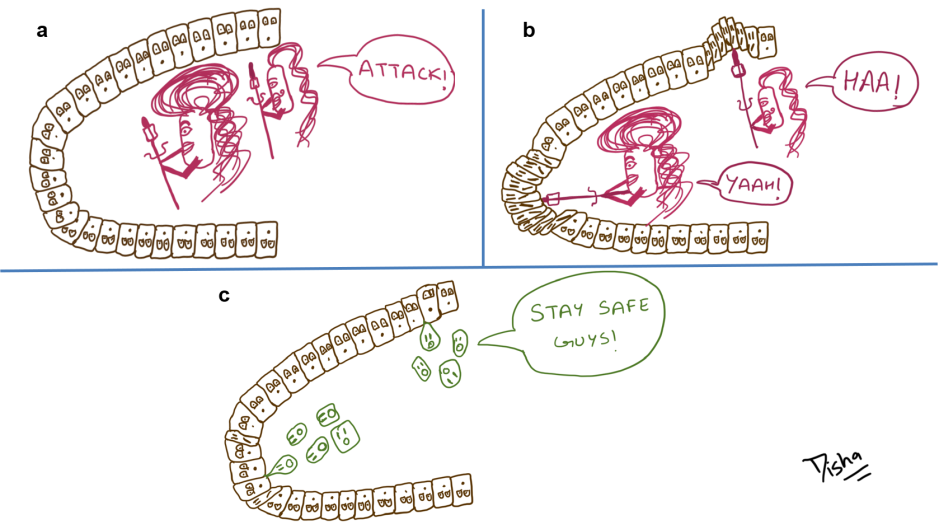
Avant de poursuivre, je dois expliquer un peu de théorie. Dans les communautés microbiennes, l’ordre dans lequel les microbes arrivent à un endroit, détermine souvent s’ils seront une espèce dominante ou s’ils vivront une vie en marge (l’effet de priorité, figure 2). La première espèce à arriver peut profiter de la plus grande quantité de nourriture, et prendra donc la plus grande partie de l’espace et/ou produira des métabolites pour inhiber la croissance des concurrentes.
Ce concept d’effet prioritaire est important, car les auteurs ont montré qu’une plante hôte peut aider sélectivement ses bactéries partenaires à surmonter l’effet prioritaire (figure 2 a droite). Wippel et Tao ont établi une corrélation entre la force d’une interaction plante-microbe et la capacité de ce même microbe à s’établir dans une communauté microbienne déjà existante (figure 3). Cela montre que les plantes peuvent “cultiver” leurs bactéries préférées.
Alors, qu’est-ce qui permet à la plante de rompre avec sa bactérie préférée ? Les chercheurs ont déterminé quels gènes étaient actifs dans les espèces végétales en utilisant la transcriptomique. Ils ont découvert que les groupes de gènes impliqués dans le système immunitaire des plantes sont activés de manière sélective par les bactéries partenaires indigènes. De même, certains gènes impliqués dans la détermination de la présence d’un ami ou d’un ennemi à l’extérieur de la racine (avec les motifs moléculaires associés aux microbes) étaient activés de manière sélective. Bien sûr, cela n’explique pas encore exactement comment la bactérie active ces gènes, mais c’est une source d’inspiration pour de nouvelles recherches.
En somme, les interactions entre les plantes et leurs partenaires bactériens sont effectivement très spécifiques et les plantes travaillent dur pour les obtenir. Mais qui de la plante ou la bactérie fait le premier clin d’œil ? Il faudra attendre un autre rencard pour cela !
Featured image: https://pickupimage.com/free-photos/Garden-beds-with-seedlings-and-leafy-greens/2332866