De la microbiologie pour ceux qui ont les crocs
L’édition de gènes au niveau supérieur
CRISPR-Cas9 a pris d’assaut le monde scientifique et a transformé l’édition du génome en une technologie précise, rapide et bon marché. Le potentiel de cette méthode semble presque illimité. Aujourd’hui, les chercheurs du laboratoire de Jennifer Doudna, qui, avec Emmanuelle Charpentier, a reçu le prix Nobel de chimie en 2020 pour le développement de la technologie CRISPR-Cas9 pour l’édition du génome, l’ont fait passer au niveau supérieur : l’édition communautaire. La recherche, publiée récemment dans Nature Microbiology, fait état de deux nouveaux outils qui permettent d’éditer le génome des microbes au sein de leurs communautés naturelles.
Les problèmes de l’édition du génome bactérien
Souvent, l’objectif des études sur le microbiome est d’identifier les bactéries clés d’une communauté microbienne qui peuvent être modifiées pour prévenir ou résoudre des maladies ou pour augmenter le rendement des cultures ou leur résistance. Pour ce faire, il faut disposer d’outils expérimentaux permettant de manipuler génétiquement des espèces bactériennes isolées dans un environnement de laboratoire. Cependant, isoler et cultiver avec succès un microbe en laboratoire peut s’avérer assez difficile. En fait, de nombreux microbes qui présentent un intérêt pour la santé humaine n’ont pas encore été cultivés avec succès en dehors de leur environnement naturel. Et les défis ne s’arrêtent pas là.
Si l’on réussit à isoler un microbe, l’étape suivante consistera à trouver le moyen optimal de modifier son génome, ce qui pourrait prendre des années ou échouer complètement. Et même si tout cela réussit, il y a de fortes chances que le microbe, une fois libéré dans sa communauté naturelle, se soit adapté à l’environnement du laboratoire et ne se comporte pas de la manière dont vous l’avez conçu.
La solution idéale pour contourner toutes ces difficultés serait de modifier les génomes bactériens dans leur environnement naturel. Et l’équipe de recherche autour de Jennifer Doudna a fourni les outils pour le faire.

Identifier la cible
Comme pour le tir d’une arme, qui nécessite de viser au préalable, les chercheurs ont commencé par sélectionner leurs cibles bactériennes au sein d’une communauté microbienne. Ils ont développé ET-seq (séquençage de transformation environnementale) pour déterminer quels microbes d’une communauté sont réellement susceptibles d’être modifiés. ET-seq utilise un transposon, qui est un élément génétique pouvant s’insérer de manière aléatoire dans les génomes.
Les chercheurs ont déterminé quelles espèces avaient incorporé le transposon en séquençant l’ADN de l’ensemble de la communauté microbienne avant et après l’ajout du transposon. Les espèces qui présentaient le plus grand nombre d’insertions étaient les plus sensibles à l’édition du génome. Grâce à ET-seq, les chercheurs ont pu cribler l’ensemble de la communauté microbienne sans avoir à isoler et à tester des espèces individuelles.
Viser juste
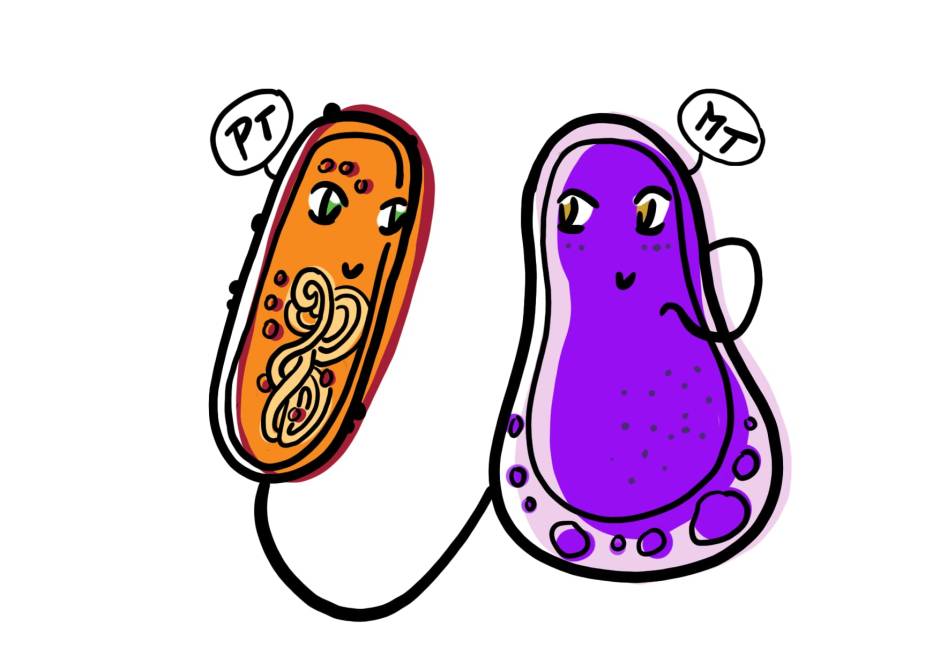
Une fois les cibles bactériennes en main, les chercheurs ont visé. Pour leur arme d’édition, ils ont mis au point un outil qu’ils ont fort justement baptisé DART (DNA-editing all-in-one RNA-guided CRISPR-Cas transposase). Comme le système CRISPR-Cas9, DART utilise une séquence d’ARN spécifique pour guider la machine d’édition vers la région génomique ciblée. Au lieu de cliver l’ADN comme le fait la protéine Cas9, DART s’appuie sur une transposase associée à CRISPR, qui insère un transposon à code-barres dans la région ciblée.
L’efficacité de ces insertions peut ensuite être facilement quantifiée à l’aide de ET-seq. “La nouvelle approche d’édition de la communauté microbienne tire parti de la technologie CRISPR et permet la modification génétique d’un gène spécifique au sein d’une bactérie spécifique”, résume Deutschbauer, l’un des auteurs de l’article de recherche.

Tirer
Pour démontrer la puissance de leurs outils, les chercheurs ont réussi à modifier des souches individuelles d’E.coli d’une communauté microbienne provenant d’un échantillon de selles d’un nourrisson. Ils ont spécifiquement ciblé des gènes qui ont été associés à des maladies.
Une arme de laboratoire seulement – pour l’instant
Cette arme puissante qui permet de modifier avec précision les gènes de bactéries spécifiques au sein de communautés microbiennes n’est encore qu’une arme de laboratoire. Le fait de contourner l’isolement ou l’ingénierie laborieuse d’espèces individuelles en laboratoire est certainement très prometteur pour les applications futures.
“À terme, nous pourrons peut-être éliminer les gènes qui causent des maladies dans vos bactéries intestinales ou rendre les plantes plus efficaces en modifiant leurs partenaires microbiens”, a déclaré Brady Cress, l’un des auteurs de l’étude. “Mais probablement, avant d’en arriver là, cette approche nous permettra de mieux comprendre comment les microbes fonctionnent au sein d’une communauté.” Car les microbes ne vivent pas en vase clos. Les étudier dans leur milieu naturel est beaucoup plus représentatif de la façon dont ils vivent et fonctionnent dans la nature.
Article original: Rubin, B.E., Diamond, S., Cress, B.F. et al. Species- and site-specific genome editing in complex bacterial communities. Nat Microbiol 7, 34–47 (2022). https://doi.org/10.1038/s41564-021-01014-7
Featured image: Unsplash and Biorender.com
Traduit par Anaïs Biclot