Breaking down the microbiology world one bite at a time
Vibrio cholerae flüchtet vor D-Aminosäuren
Bakterien leben, genau wie wir, in Gemeinschaften, in denen sie mit ihren Nachbarn interagieren müssen. Wenn es in ihrer Umgebung nur begrenzte Ressourcen gibt, konkurrieren die Bakterien miteinander. Während solcher Nachbarschaftskämpfe setzen Bakterien giftige Proteine in ihre Umgebung frei, um die feindlichen Bakterien zu töten. Eine der angewandten Strategien besteht beispielsweise darin, freie Aminosäuren abzugeben.
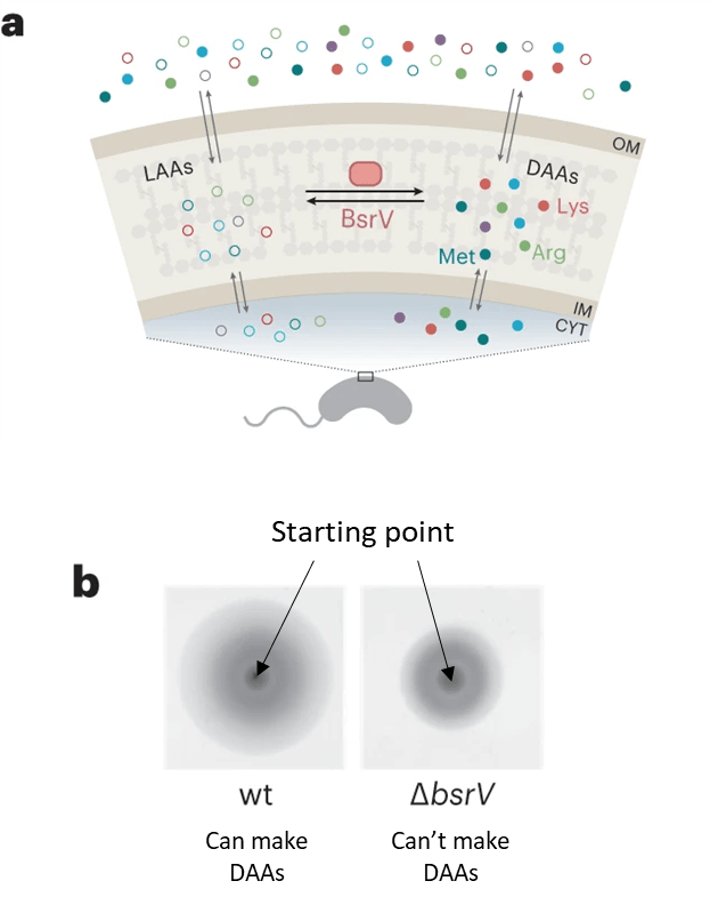
Proteine bestehen aus Aminosäuren, das ist gemeinhin bekannt. Viele wissen jedoch nicht, dass nicht alle Arten von Aminosäuren zum Aufbau von Proteinen verwendet werden. Es gibt nämlich zwei Arten von Aminosäuren: L-Aminosäuren (LAAs) und D-Aminosäuren (DAAs). Diese beiden Typen haben zwar die gleiche Zusammensetzung, ihre chemischen Strukturen sind jedoch spiegelbildlich zueinander aufgebaut, wie unten dargestellt. Während die meisten Lebensformen auf der Erde LAAs zur Bildung von Proteinen verwenden, können Bakterien LAAs in DAAs umwandeln. DAAs werden von Bakterien als Toxine verwendet, um ihre feindlichen Nachbarn zu töten. Man weiß jedoch nicht genau, wie sich die DAAs auf das freisetzende Bakterium selbst auswirken.

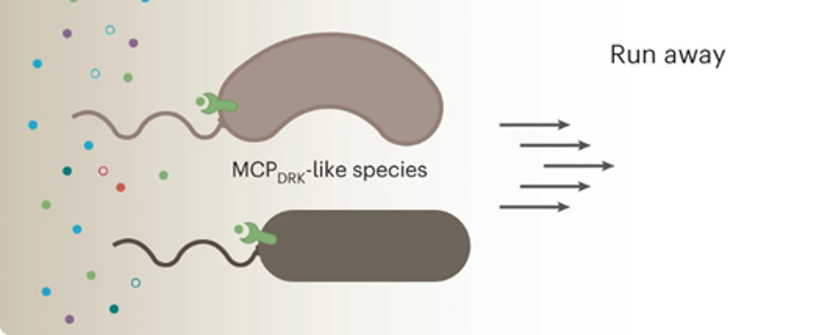
Die Wissenschaftler*innen der hier beschriebenen Studie verwendeten Vibrio cholerae, um die Auswirkungen von DAAs auf das Verhalten von Bakterien zu verstehen. Vibrio cholerae ist ein Bakterium, das beim Menschen die Krankheit Cholera verursacht und dafür bekannt ist, dass es eine hohe Konzentration an DAAs in seine Umgebung abgibt. Die Forschenden fragten sich, welche Defekte ein Bakterium wohl haben würde, wenn es keine DAAs produzieren könnte. Es ist bereits bekannt, dass Vibrio ein Protein namens BsrV nutzt, um LAAs in DAAs umzuwandeln (siehe Abbildung unten). Die Forschenden untersuchten also die Defekte in Vibrio cholerae Bakterien, die kein BsrV mehr hatten. Sie stellten fest, dass diese Bakterien sich schwimmend nicht so weit vom Startpunkt entfernten wie die Bakterien, die noch DAAs produzieren konnten (siehe Abbildung unten). Dies könnte bedeuten, dass Bakterien, die keine DAAs produzieren, sich nicht richtig bewegen können. Eine andere Erklärung wäre, dass die Bakterien unter normalen, DAA-produzierenden Umständen vor dem Ort flüchten, an dem sie große Mengen DAAs freigesetzt haben.
Bakterien besitzen fadenförmige Strukturen, die Geißeln, mit denen sie sich fortbewegen. Die Wissenschaftler*innen überprüften daher, ob die Bakterien, die keine DAAs produzieren konnten, noch funktionelle Geißeln besaßen. Erstaunlicherweise waren die Geißeln tatsächlich funktionstüchtig. Das machte die Forschenden neugierig, ob Bakterien stattdessen die DAAs wahrnehmen und vor diesen flüchten konnten. Durch die Freisetzung von DAAs in die Umgebung könnten die Bakterien dann nicht nur ihre Nachbarn töten, sondern auch stressige Bedingungen wie Hunger und ungünstige pH- oder Temperaturwerte vermeiden.
Bakterien können insgesamt 20 verschiedene DAAs produzieren. Um herauszufinden, welche davon die Beweglichkeit von Bakterien beeinflussen, führten die Forschenden sogenannte Chemotaxis-Tests durch. Damit fanden sie heraus, dass Bakterien nur zwei der DAAs, nämlich D-Arginin und D-Lysin, wahrnehmen und vor beiden flüchten konnten. Mit der hochmodernen Technik des thermalen Proteom-Profilings fanden sie anschließend den Rezeptor, den die Bakterien für die Erkennung der DAAs nutzten. Diese Technik beruht auf dem Prinzip, dass Rezeptoren bei Kontakt mit ihrem Liganden hitzestabiler werden. So konnten die Forschenden identifizieren, dass Bakterien D-Arginin über ein Rezeptorprotein namens VC1313 oder MCPDRK wahrnahmen.
Da der reine Vorgang des Erkennens durch einen Rezeptor eine Menge Energie erfordert, versuchten die Forschenden, den genauen Zeitpunkt zu identifizieren, an dem die Bakterien die DAAs wahrnahmen. Sie markierten den MCPDRK-Rezeptor mit einem grün fluoreszierenden Protein (GFP) und konnten so beobachten, dass die Bakterien MCPDRK produzierten, sobald sie in die sogenannte stationäre Phase eintraten (siehe Abbildung unten). Dies ist ein Stadium des Bakterienwachstums, in dem Nährstoffe nur noch begrenzt verfügbar sind und die Umgebung das Wachstum der Bakterien nicht mehr optimal fördert. Später zeigten sie außerdem, dass die Produktion von MCPDRK von RpoS abhängt. Das ist ein Faktor, der die Produktion bestimmter Proteine steuert, sobald Bakterien in die stationäre Phase eintreten oder einer Stresssituation ausgesetzt sind.

In diesem Artikel wurde also zusammenfassend gezeigt, dass Bakterien dazu in der Lage sind, vorhandene DAAs zu erkennen, wenn sie ungünstige Bedingungen vorfinden. Unter diesen Umständen flüchten die Bakterien dann vor einem Ort mit zu vielen DAAs (siehe letzte Abbildung), auf der Suche nach anderen, günstigeren Lebensbedingungen in ihrer Umgebung.
Bakterielle Motilität steht in einem gegensätzlichen Verhältnis zur Biofilmbildung. Bakterien in einem Biofilmzustand sind resistent gegen Antibiotika und daher eine der Hauptursachen für Todesfälle durch krankenhausbedingte Infektionen. Die vorliegende Studie gibt uns nun einen Hinweis darauf, was Bakterien dazu veranlasst, sich zu bewegen. Eine andere Studie hat außerdem gezeigt, dass die Aktivierung einiger Gene, wie bsrV, die Biofilmbildung behindert. Nun stellt sich die Frage, ob DAAs sozusagen der Schalter zwischen einem antibiotikaresistenten Biofilmzustand und einem antibiotikaempfindlichen, mobilen Zustand sind? Wenn wir die Biologie hinter diesem Umschaltmechanismus noch besser verstehen und erforschen, dann könnte dies in der Zukunft zur Entwicklung besserer Therapeutika beitragen.

Link to the original post:
“D-amino acids signal a stress-dependent run-away response in Vibrio cholerae.”Irazoki O, Ter Beek J, Alvarez L, Mateus A, Colin R, Typas A, Savitski MM, Sourjik V, Berntsson RP, Cava F. Nature Microbiology, 2023 Aug. 10.1038/s41564-023-01419-6
Featured image: Image adapted from the original article Irazoki et al