Deconstruyendo la microbiología mordisco a mordisco
Moviéndose a través de las fronteras: cómo los genes bacterianos se vuelven móviles
Las bacterias llevan diferentes “manuales” o “guías” con instrucciones codificadas dentro de su ADN. Estos manuales permiten, entre otras cosas, construir enzimas u otras proteínas que posibilitan el movimiento de ADN dentro de su propio genoma o entre diferentes bacterias [1]. Esos manuales de construcción también se denominan Elementos Genéticos Móviles (MGEs por sus siglas en inglés) e incluyen, por ejemplo, plásmidos, bacteriófagos y transposones. Mientras que los plásmidos se organizan en entidades autorreplicantes estables o “replicones” dentro de las bacterias, los bacteriófagos pueden inyectar su genoma e integrarlo en la célula bacteriana y el cromosoma. Por otro lado, los transposones pueden “saltar” aleatoriamente al ADN del fago o del plásmido, lo que potencialmente cambia las propiedades de la célula infectada [1]. Todos estos mecanismos permiten que ciertos genes se muevan de una célula bacteriana a la siguiente y, en mayor escala, viajen de un hábitat como el cuerpo humano al siguiente, por ejemplo, animales o el medio ambiente.
La investigación sugirió la presencia de Genes de Resistencia a los Antibióticos (ARGs por sus siglas en inglés) dentro de los MGE. Carbapenem es una clase de antibióticos con un amplio espectro de actividad antimicrobiana contra bacterias grampositivas y gramnegativas. Un mecanismo por el cual estas bacterias ganan resistencia contra carbapenem es la expresión de carbapenemasas. Las carbapenemasas pertenecen al grupo de hidrolasas que hidrolizan y por lo tanto inactivan a los antibióticos de la clase Carbapenem. Los genes que codifican carbapenemasas, como blaOXA-48, se encuentran en plásmidos de Klebsiella pneumoniae, donde son responsables de la expresión de carbapenemasas [2]. Las enterobacterias resistentes a los carbapenémicos son un grupo de bacterias que incluyen a Klebsiella pneumoniae, Acinetobacter baumannii o Pseudomonas aeruginosa, que comúnmente causan enfermedades infecciosas en entornos de atención médica, por ejemplo hospitales, pero para los cuales falla el tratamiento con antibióticos carbapenémicos [3]. Si bien este grupo de bacterias ha sido ampliamente estudiado en humanos, se sabe poco sobre la epidemiología y transmisión de éstas en el medio ambiente o en los animales.
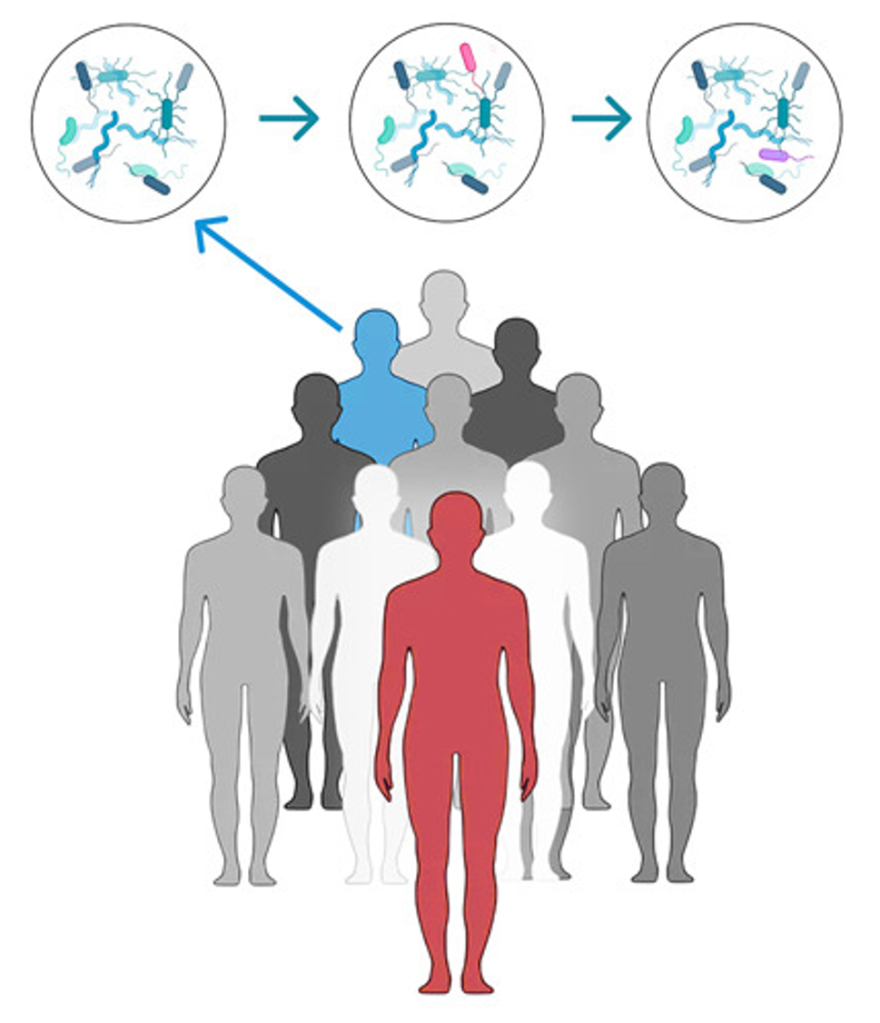
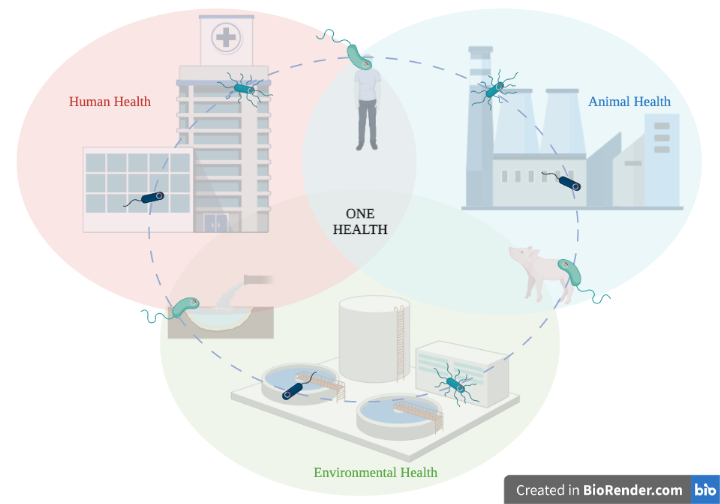
Los seres humanos están constantemente en contacto con las bacterias a través de su entorno, por ejemplo, al tocar superficies, plantas, animales o al comer y beber. Todas estas son rutas de viaje para que las bacterias y sus genes de resistencia a los antibióticos crucen las fronteras humanas y se propaguen no solo a nivel local sino también global. Por lo tanto, los investigadores y las organizaciones de salud ahora se están enfocando en el llamado enfoque de “Una sola salud” (“One Health” en inglés) para combatir la propagación de la resistencia a los antimicrobianos, que se enfoca en la acción colaborativa entre los sectores humano, ambiental y animal [4] (Figura 1).
Ramsamy y sus colegas estudiaron y compararon MGEs de Enterobacterias resistentes a los carbapenemes en humanos, el medio ambiente y los animales dentro de la misma área geográfica en Sudáfrica utilizando un enfoque de “Una sola salud” [5]. Los investigadores pudieron aislar diecinueve Enterobacteriales productores de carbapenemasas de tres hospitales del sector público y cuerpos de agua de una planta de tratamiento de aguas residuales, y encontraron una alta prevalencia de K. penumoniae (47%) y Enterobacter hormaechei (32%). Sin embargo, no encontraron cepas bacterianas aisladas en cerdos de una planta de procesamiento de alimentos que incluía un matadero.
Posteriormente, caracterizaron las cepas aisladas como multirresistentes a antibióticos como ceftazidima, cefuroxima y ertapenem. Además, dieciséis de diecinueve cepas aisladas que eran resistentes a al menos un carbapenem también contenían genes que codifican carbapenemasas con la prevalencia más alta para blaOXA-181 y blaNDM-1. Ambos genes fueron comunes en cepas bacterianas aisladas de humanos y del medio ambiente.
En total, el grupo de investigación identificó cuarenta y dos plásmidos de las diecinueve cepas aisladas. Dentro de los plásmidos, encontraron ARG junto con elementos transponibles y/o secuencias de inserción. Por ejemplo, una mirada más cercana al entorno genético que rodea al gen blaOXA-181 que codifica la carbapenemasa reveló la presencia del gen ereA. Este último codifica para la eritromicina esterasa, que transmite una alta resistencia al antibiótico eritromicina. Además, también se encontraron secuencias de inserción como IS3000 y transposones como Tn3 en plásmidos que albergan blaOXA-181. Por lo tanto, uno puede imaginar a los MGE como un vehículo que incorpora varios genes de resistencia a los antibióticos y se mueve entre las células bacterianas hacia nuevos sectores.
Sin embargo, los investigadores también señalaron los límites de este estudio en términos de generalización debido al pequeño número de cepas bacterianas aisladas. Para confirmar los conocimientos adquiridos sobre la propagación de la resistencia a los antimicrobianos en un contexto de “Una sola salud”, sugirieron más investigación con tamaños de muestra más grandes y en diferentes ubicaciones geográficas.
Actualmente, se han encontrado genes de resistencia a los antibióticos en MGE en suelos del Ártico alto [6], así como en agua potable en la India [7]. Esto indica la propagación de gran alcance de la resistencia a los antibióticos a diferentes sectores en todo el mundo y sugiere MGE como impulsor. Sin embargo, los MGE pueden contener no sólo genes de resistencia a los antibióticos, sino también genes de virulencia u otros genes de resistencia, lo que implica más consecuencias en la salud humana para las enfermedades infecciosas bacterianas existentes [3].
Link to the original post: Ramsamy, Y., et al. (2021). “Mobile genetic elements-mediated Enterobacterales-associated carbapenemase antibiotic resistance genes propagation between the environment and humans: A One Health South African study.” Sci Total Environ 806(Pt 3): 150641.
Featured image: Photo by Norbert Dudas on Unsplash
References:
- Frost, L. S., et al. (2005). “Mobile genetic elements: the agents of open source evolution.” Nat Rev Microbiol 3(9): 722-732.
- Yang, X., et al. (2021). “Carbapenem Resistance-Encoding and Virulence-Encoding Conjugative Plasmids in Klebsiella pneumoniae.” Trends Microbiol 29(1): 65-83.
- CDC – Carbapenem-resistant Enterobacterales (CRE) – https://www.cdc.gov/hai/organisms/cre/index.html November 9, 2021
- WHO – WHO,FAO, and OIE unite in the fight against Antimicrobial Resistance – https://www.who.int/foodsafety/areas_work/antimicrobial-resistance/amr_tripartite_flyer.pdf November 9, 2021
- Ramsamy, Y., et al. (2021). “Mobile genetic elements-mediated Enterobacterales-associated carbapenemase antibiotic resistance genes propagation between the environment and humans: A One Health South African study.” Sci Total Environ 806(Pt 3): 150641.
- McCann, C. M., et al. (2019). “Understanding drivers of antibiotic resistance genes in High Arctic soil ecosystems.” Environ Int 125: 497-504.
- Walsh, T. R., et al. (2011). “Dissemination of NDM-1 positive bacteria in the New Delhi environment and its implications for human health: an environmental point prevalence study.” Lancet Infect Dis 11(5): 355-362.
Traducido por: Julián E. Prieto-Vivas