Microbiologie in hapklare porties
CRISPRi: een DNA-demper die de moeite waard is om lawaai over te maken
Wat is CRISPRi en hoe werkt het?
CRISPR is een krachtig genbewerkingstool waarmee wetenschappers DNA-sequenties in cellen met hoge precisie kunnen wijzigen (zie ons vorige artikel over het identificeren van doelwitten). Het is een waardevol hulpmiddel voor onderzoek en therapeutische toepassingen. Het maakt gebruik van een systeem dat uit twee delen bestaat: (i) een eiwitenzym genaamd Cas9 dat DNA kan binden en knippen, en (ii) een RNA-molecuul genaamd sgRNA, dat Cas9 naar een specifieke DNA-sequentie leidt (lees meer over het CRISPR-Cas systeem).
CRISPRi (interferentie) is een aangepaste versie van dit gereedschap waarbij het enzym Cas9 is geïnactiveerd. Het kreeg de naam dCas9, voor “dood” Cas9. Dit inactieve enzym kan nog steeds DNA binden, maar het kan geen DNA knippen. Wanneer dCas9 aan DNA bindt, blokkeert het DNA-transcriptie, waardoor genexpressie nauwkeurig kan worden geregeld zonder de DNA-sequentie te wijzigen. Omdat dCas9 DNA-transcriptie blokkeert, wordt gezegd dat het genexpressie “onderdrukt”. Door een specifieke chemische stof toe te voegen die een “inducer” wordt genoemd, kunnen wetenschappers het CRISPRi-systeem naar believen “aan” en “uit” zetten, waardoor een extra controlelaag wordt toegevoegd. Het systeem kan zelfs worden gebruikt om de expressie van meerdere genen tegelijkertijd te controleren (multiplexing)!
CRISPRi-technologie wordt al gebruikt in biotechnologie, onderzoek en therapeutica, waaronder:
Metabolische engineering: om metabolische routes in cellen om te leiden om specifieke verbindingen te produceren, zoals biobrandstoffen of geneesmiddelen
Ziekte onderzoek: om de rol van genen in de ontwikkeling van ziekten te bestuderen, wat kan leiden tot de identificatie van nieuwe doelwitten voor geneesmiddelen.
Synthetische biologie: genetische circuits bouwen die gebruikt kunnen worden om het gedrag van cellen te sturen, zoals de activering van specifieke pathways in reactie op signalen uit de omgeving.
Wat zijn de barrières?
Belangrijke factoren in het ontwerp van het CRISPRi-systeem bepalen de efficiëntie van de genonderdrukking; hoeveel de expressie van het gen kan worden voorkomen. Eerdere onderzoekers hebben geavanceerde, maar enigszins beperkte methoden gebruikt om de efficiëntie van genonderdrukking te controleren.
Voorbeelden zijn:
- het veranderen van het expressieniveau van dCas9 of het sgRNA met verschillende promotorsterktes (initiator van DNA-transcriptie)
- veranderen van de concentratie van de “inducer
- variëren van het kopiegetal van het plasmide van waaruit het CRISPRi-systeem tot expressie wordt gebracht
- wijzigen van de sgRNA-doelpositie
- gebruik van een ander gastheer micro-organisme
Deze methoden hebben echter een aantal nadelen. Het meest opvallende is dat de ontwerpelementen die hierboven genoemd zijn, aangepast moeten worden voor elk nieuw gen dat getarget wordt om de gewenste efficiëntie van genonderdrukking te bereiken. Het testen van verschillende ontwerpelementen is tijdrovend en kostbaar omdat onze huidige kennis het niet mogelijk maakt om voorspelbare, reproduceerbare wijzigingen aan te brengen. In plaats daarvan is trial and error vaak nodig.
In een nieuw onderzoek heeft de onderzoeksgroep veel van deze problemen opgelost door structurele elementen van het sgRNA bloot te leggen die voorspelbare niveaus van onderdrukking kunnen bieden, ongeacht het doelgen. Laten we eens kijken hoe ze dat hebben gedaan.
Verbeteren van sgRNA-ontwerpen
Eerst ontwierp de groep een snelle en eenvoudige manier om te meten hoe effectief hun systeem het doelgen onderdrukte. Ze integreerden het gen voor een fluorescerend eiwit (mCherry) in het genoom van E. coli op een plaats waar het constitutief (constant) tot expressie zou komen. Vervolgens ontwierpen ze een bibliotheek van verschillende sgRNA’s met verschillende sequenties voor het doelgen. Met het fluorescerende eiwit konden ze visualiseren welke sgRNA’s het meest effectief waren in het onderdrukken van het gen – hoe minder fluorescentie, hoe effectiever de onderdrukking.
De resultaten toonden aan dat het wijzigen van de sgRNA-sequentie in een gebied genaamd de tetraloop (en de flankerende sequenties) een set sgRNA’s opleverde met verschillende onderdrukkingsefficiënties. De echte game-changer is dat toen ze de doelsequentie van het sgRNA veranderden naar andere genen, de modificaties nog steeds voorspelbare niveaus van onderdrukking opleverden (tot 45-voudig). Niet alleen dat, maar ze observeerden ook minder off-target effecten (veranderingen in de expressie van niet gerelateerde genen) en ook geen toxiciteit van zowel de “inducer” of de expressie van dCas9. Hoewel hun systeem nog steeds enigszins gevoelig was voor de concentratie van de toegevoegde “inducer” en de sterkte van de gebruikte dCas9-promotor, was het gevoeligheidsniveau een enorme verbetering ten opzichte van de eerder beschreven systemen.
Om beter te begrijpen hoe de verschillende sgRNA tetraloop-sequenties leidden tot verschillende niveaus van onderdrukking, gebruikte de groep een computationele benadering die de verandering in vrije energie meet wanneer het sgRNA en dCas9 op elkaar inwerken. De groep analyseerde hun bibliotheek van sgRNA’s met deze methode en ontdekte dat de vrije energieverandering correleerde met het niveau van onderdrukking (en vermoedelijk de stabiliteit van de interactie tussen het sgRNA en dCas9). De volgende stap was om aan te tonen dat de ontdekking kon worden toegepast op een praktijkvoorbeeld.
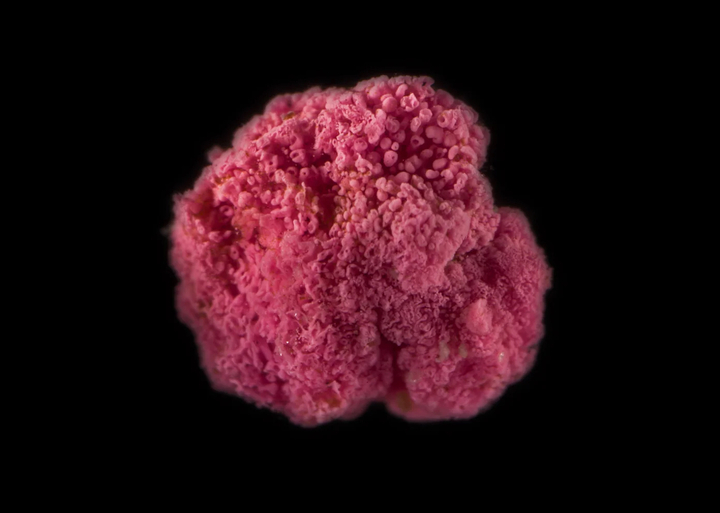
De biosynthese van lycopeen in E. coli verfijnen
Als proof of concept koos de groep ervoor om hun nieuwe CRIPSRi-technologie toe te passen om de biosynthese van lycopeen te verbeteren. Lycopeen is een natuurlijk rood pigment in de familie van carotenoïde verbindingen. Het is het pigment dat vruchten zoals tomaten en watermeloenen hun kenmerkende rode kleuren geeft. Lycopeen is een waardevolle verbinding vanwege zijn antioxiderende eigenschappen die voordelen voor de gezondheid kunnen hebben. Synthese van lycopeen is een ingewikkelde, multi-enzymroute die begint met eenvoudige precursoren (zoals pyruvaat en glyceraldehyde-3-fosfaat).
Deze precursoren worden ook gebruikt in het centrale koolstofmetabolisme van bacteriële cellen en zijn dus veelgevraagd. Dit betekent dat als het CRISPRi-systeem wordt gebruikt om te veel van de precursormoleculen weg te leiden van het centrale koolstofmetabolisme en in de richting van lycopeenproductie, de cellen zullen afsterven. Er is een zorgvuldige balans nodig! Dit was het perfecte voorbeeld om de exquise fijnafstemming van de genexpressie aan te tonen die mogelijk is met het nieuwe CRISPRi systeem. De groep ontwierp 5 verschillende sgRNA’s met verschillende niveaus van onderdrukking, gericht op het enzym dat bepaalt of de precursormoleculen terechtkomen in het centrale koolstofmetabolisme of in de lycopeenproductie. Vervolgens vergeleken ze hun systeem met een traditioneler CRISPRi-systeem dat niet in staat was om de benodigde regulatie te bereiken en de cellen doodde. Hun systeem verbeterde echter de lycopeenproductie aanzienlijk (2,7-voudige toename) en vertoonde geen groeistoornissen.
Het nieuwe CRISPRi-systeem werd gebruikt om genexpressie op een voorspelbare, robuuste manier te controleren, ongeacht het doelgen. Het bleek effectief te zijn in het manipuleren van bacteriële metabolische flux om de productie van een zeer waardevolle verbinding te verbeteren: lycopeen. Dit nieuwe hulpmiddel en toekomstige ontwikkelingen in de CRISPRi-technologie zullen waarschijnlijk belangrijk zijn voor mensen om met micro-organismen te werken en samen de grenzen van de biotechnologie te verleggen.
Link to the original post: Gibyuck Byun, Jina Yang, Sang Woo Seo. CRISPRi-mediated tunable control of gene expression level with engineered single-guide RNA in Escherichia coli, Nucleic Acids Research, 2023. https://doi.org/10.1093/nar/gkad234.
Featured image: Made by author
Vertaald door: Liang Hobma