Microbiologie in hapklare porties
Technologie om virale infecties aan te pakken
Oxford Nanopore sequencing is een technologie die gebruikt kan worden om genetisch materiaal (DNA of RNA) van virussen af te lezen. Deze technologie kan helpen bij het monitoren van virusinfecties omdat het snel en nauwkeurig het specifieke type virus kan identificeren dat de infectie veroorzaakt.
Traditioneel worden virale infecties gecontroleerd door een monster van een patiënt te nemen en vervolgens verschillende methoden te gebruiken om het virus te identificeren (zoals een PCR). Deze methoden kunnen echter tijdrovend zijn en niet altijd nauwkeurige resultaten opleveren. Oxford Nanopore sequencing daarentegen kan snelle en nauwkeurige resultaten opleveren, waardoor het een nuttig hulpmiddel is voor het monitoren van virale infecties.
Deze methode is snel en nauwkeurig omdat het een real-time sequencing technologie is die direct het DNA of RNA molecuul leest terwijl het door een kleine porie of kanaal passeert.
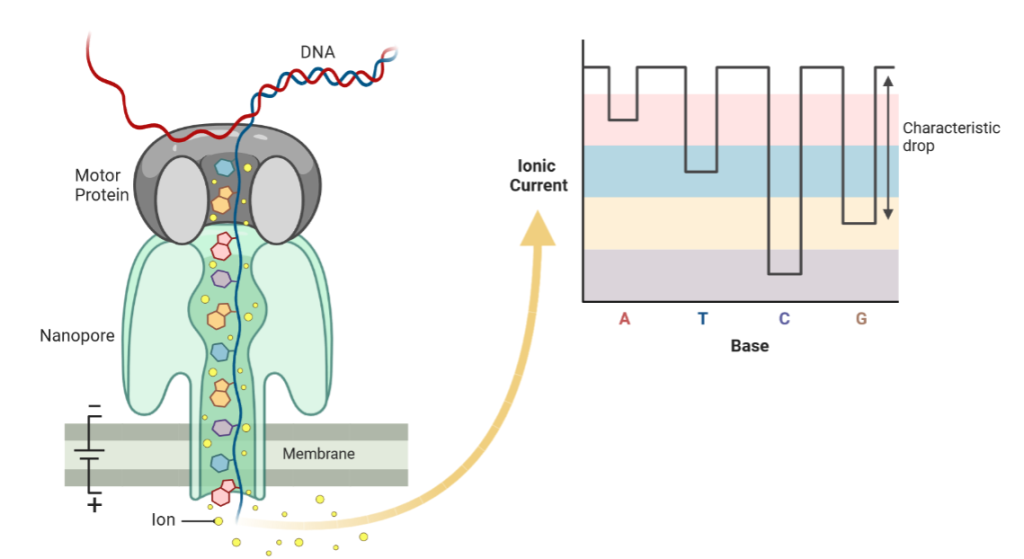
Het DNA- of RNA-molecuul is elektrisch geladen en als het door de porie gaat, veroorzaakt het veranderingen in de elektrische stroom, die in realtime gedetecteerd en geanalyseerd kunnen worden. Omdat Oxford Nanopore sequencing direct het DNA of RNA molecuul leest, kan het bovendien langere lees lengtes en een hogere mate van nauwkeurigheid bieden in vergelijking met andere sequencing technologieën. Dit komt doordat andere sequentiemethoden mogelijk amplificatie of fragmentatie van het DNA of RNA vereisen, wat fouten of vertekeningen kan introduceren in de resulterende sequentie gegevens.
Maar hoe kunnen we het gebruiken om virale infecties te monitoren?
In hun onderzoek ontwikkelden Munroe en collega’s een methode om het genetisch materiaal van virussen, zoals het SARS-CoV-2 virus dat verantwoordelijk is voor de COVID-19 pandemie, efficiënter en kosteneffectiever te sequentiëren. Veel verschillende laboratoria hebben gekeken naar het gebruik van nanopore sequencing om virale infecties zelfs bij dieren aan te pakken om boeren een snellere en nauwkeurigere diagnose te kunnen bieden.
Een uitdaging bij het gebruik van deze sequencingtechnologie is het verkrijgen van voldoende DNA of RNA om de machine nauwkeurig te laten werken. In dit onderzoek ontwikkelden de auteurs een experimentele procedure om te voorspellen wanneer er voldoende DNA of RNA-gegevens werden verkregen uit elk monster voor genetische gegevens van hoge kwaliteit. Om de efficiëntie van Oxford Nanopore sequencing verder te verbeteren, ontwikkelden de onderzoekers een tool die adaptieve sampling op de sequencing machine automatiseert tijdens het sequencing proces. Adaptive sampling is een manier om specifieke moleculen te selecteren en te sequencen voor analyse. In plaats van traditionele methoden te gebruiken om een interessante DNA- of RNA-sequentie te targeten, zoals depletie of verrijking van de sequentie in een lab, heeft Nanopore een software gestuurde aanpak ontwikkeld. Dit stelt onderzoekers in staat om individuele poriën te controleren en te verzoeken om het uitwerpen van een specifieke molecuul die in een porie wordt gesequenced. Met andere woorden, gebruikers kunnen op verzoek stoppen met het verzamelen van de sequentie gegevens van een DNA- of RNA-molecuul. Dit helpt bij het balanceren van de hoeveelheid sequencing dekking over verschillende monsters en regio’s van het genetisch materiaal van het virus, waardoor de tijd die nodig is om volledige genetische sequenties van het virus te verkrijgen, wordt verkort.
Met andere woorden, adaptieve bemonstering maakt het testen op het SARS-CoV-2 virus op een snellere en efficiëntere manier mogelijk omdat de SARS-CoV-2 sequenties direct kunnen worden gericht. Door gebruik te maken van deze methode was het team in staat om de sequenties van de virale genomen in een kortere tijd te verkrijgen en de monsters alleen uit te voeren als dat nodig was, waardoor een snellere omzet mogelijk was. Terwijl ze tijd besparen, toonden de auteurs aan dat dit geen invloed heeft op de nauwkeurigheid van de resultaten. Op dit moment kunnen 96 monsters worden getest met deze methode, en in de toekomst kunnen we er misschien nog meer testen!
Over het algemeen zijn deze verbeteringen in de efficiëntie en nauwkeurigheid van sequencing belangrijk voor het monitoren en bewaken van virale pathogenen, maar ook voor het ontwikkelen van behandelingen en vaccins om infectieziekten zoals COVID-19 te bestrijden.
Featured image: made with biorender template
Vertaald door: Liang Hobma