Deconstruyendo la microbiología mordisco a mordisco
¿Cómo perciben su microbioma las plantas?
Muchos artículos en este blog muestran, los microbios juegan diversos roles en ecosistemas dependiendo el contexto. Por ejemplo, pueden ser usados para elaborar queso o balanceando el microbioma intestinal, pero también pueden ser peligrosos cuando infectan pacientes que reciben trasplantes de medula. Entonces, como los organismos hospedadores pueden determinar ¿cuándo atacar o acoger a microbios que pueden colonizarlos? Investigadores en Zúrich se propusieron abordar esta pregunta investigando cómo las plantas perciben su microbioma. Para ello, los investigadores miraron la planta modelo Arabidopsis thaliana, la cual es usada extensivamente en investigación con la finalidad de extrapolar los resultados.
El equipo identifico un grupo de genes en A. thaliana que son consistentemente activados cuando bacterias colonizan las hojas. Plantearon la hipótesis de que estos genes de “respuesta general a lo no propio” (GNSR, por sus siglas en inglés) constituyen una estrategia de adaptación defensiva que protege contra patógenos. Además, la expresión de estos genes se correlaciona con la cantidad de microbios que colonizan la planta: mayor expresión de los genes a mayor cantidad de microbios.
En un estudio reciente siguiendo esta línea de investigación, los investigadores estudiaron los genes de GNSR con relación a microbios no patogénicos. comparte sus ideas sobre la inmunidad vegetal y su trabajo en un laboratorio de microbiología vegetal:
Percibir lo ajeno
“Estos genes de GNSR no eran un descubrimiento reciente”, explica Andreas, “pero agruparlos como una respuesta consistente a señales ajenas fue una revelación. La idea es que la planta percibe señales ajenas, ya sean de patógenos o comensales, y existe una respuesta basal a esta percepción que incluye estos genes. El primer estudio se centró en las interacciones patógenas, mientras que el trabajo más reciente explora cómo la GNSR afecta a la microbiota no patógena.”
La importancia de investigar por cuenta propia
Al principio del proyecto, el equipo se topó con cierta confusión sobre si la GNSR representaba una respuesta inmunitaria realmente novedosa.
“Acababa de empezar mi doctorado cuando me uní al proyecto”, recuerda Andreas. “Nuestro laboratorio había estado en contacto con colaboradores que habían identificado conjuntos de genes en Arabidopsis que se activan mediante diversas moléculas bacterianas. Creíamos haber corroborado la coincidencia entre estos genes de Arabidopsis y los nuestros, pero al parecer, nuestros colaboradores habían malinterpretado esa comparación.”
Como resultado, a Andreas se le encomendó la tarea de investigar las diferencias entre los la GNSR y las respuestas inmunitarias descritas anteriormente. “Dedicamos mucho tiempo a intentar comprender esta supuesta diferencia, pero nunca llegó a encajar del todo. Aun así, era nuevo en el laboratorio y simplemente confiaba en que el trabajo preliminar ya estaba hecho.”
Finalmente, Andreas decidió revisar los datos y encontró una coincidencia significativa entre la GNSR y las respuestas inmunitarias conocidas. En lugar de verlo como un contratiempo, Andreas lo consideró parte del proceso científico.
“Esto me hizo comprender la importancia de verificar la información y realizar análisis propios, incluso si se cree que ya se han realizado.”
Experimentos cooperativos
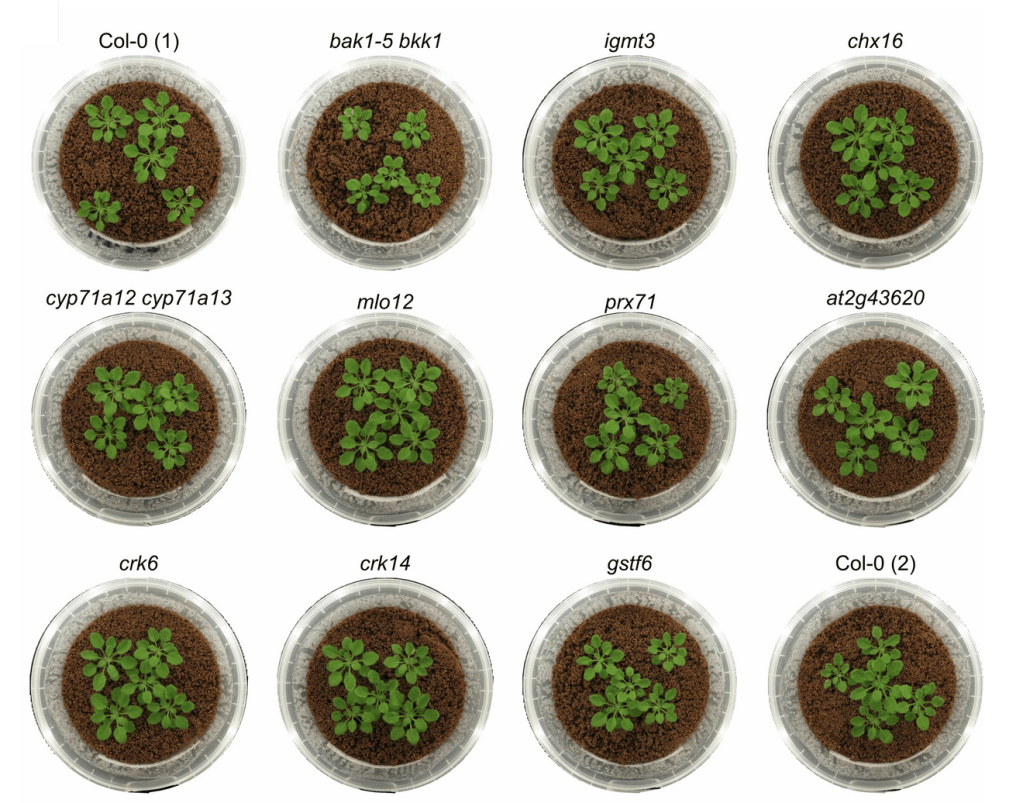
Para estudiar las interacciones entre Arabidopsis thaliana, los genes de GNSR y el microbioma, los investigadores cultivaron miles de pequeñas plantas en el laboratorio.
“Contamos con excelentes sistemas de cultivo para cultivar plantas en condiciones estériles e inocularlas con bacterias foliares específicas o comunidades bacterianas definidas. También utilizamos diferentes líneas de plantas mutantes, todo en un entorno muy controlado. Dado que los sistemas biológicos son complejos, se necesita una gran cantidad de plantas para extraer conclusiones de los análisis estadísticos. Y como las plantas tardan en crecer y no se sabe qué ocurrirá en el experimento, también realizamos muchos experimentos en paralelo. Durante la revisión del artículo, cultivamos, inoculamos y cosechamos alrededor de 2500 plantas.”
Por suerte, Andreas no tuvo que trabajar solo. “Siempre colaboramos: cuando cosechamos plantas para experimentos donde probamos muchas condiciones en paralelo, dos personas cortan las plantas, dos procesan las muestras y una realiza mediciones. Esto nos lleva prácticamente todo el día para unas 400 a 600 plantas. ¡El laboratorio es un lugar fantástico para trabajar porque colaboramos constantemente!”
Comprender la salud de las plantas
Comprender cómo interactúan las plantas sanas con su microbioma es fundamental para el futuro de la agricultura. Andreas explica que gran parte del conocimiento sobre las interacciones planta-bacteria proviene de estudios sobre patógenos y cómo causan enfermedades. “Esta investigación fue crucial y nos ha traído hasta aquí; nos encontramos en un punto en el que nos damos cuenta de que esto es solo una parte de la historia. Para diseñar microbiomas para su aplicación en la agricultura, necesitamos la otra mitad: comprender qué constituye un sistema sano. Estamos intentando caracterizar a nivel molecular cómo es un sistema sano.”
Se necesita una validación rigurosa
Andreas invita a investigadores de otros laboratorios a repetir los experimentos y asegurarse de que sus hallazgos se mantengan en diversas circunstancias. “Es genial ver que este estudio ha atraído más atención de la que esperaba, pero espero que también genere críticas constructivas. De hecho, un artículo publicado unos meses después del nuestro reprodujo uno de nuestros resultados, ¡y eso fue muy alentador! Estos sistemas tienden a ser muy complejos y un tanto artificiales, así que es importante contar con una validación rigurosa.”
Enlace al artículo original: Keppler, A., Roulier, M., Pfeilmeier, S. et al. Plant microbiota feedbacks through dose-responsive expression of general non-self response genes. Nat. Plants 11, 74–89 (2025).
Imagen destacada: Keppler et al. 2025